Numcraw for R User
Tomorrow, Japan.R will be held.
It is one of the biggest R user meetup.
If you don't know about it, I recommend you to google it.
Unfortunately, I can not join Japan.R because of a business trip.
So following slide is to celebrate Japan.R from U.S.
The function Numacraw() in the slide is here.
numacraw<-function(get2=FALSE){
if(require(png) && require(RCurl)){
set.seed(Sys.time())
path_back<-paste0(tempdir(),"\\back.png")
download.file("http://pbs.twimg.com/media/B4AEO4LCEAMjpx7.png"
, destfile = path_back,mode="wb")
path_front<-paste0(tempdir(),"\\front.png")
download.file("http://pbs.twimg.com/media/B4AB8oDCIAA_04r.png"
, destfile = path_front,mode="wb")
vec<-par()$usr
SIZE<-runif(min=0.4,max=1, 1)
X<-runif(min=0, max=1, 1)*(vec[2]-vec[1])
Y<-runif(min=0, max=1, 1)*(vec[4]-vec[3])
D<-ifelse(runif(min = 0 ,1)>0.3, path_front, path_back)
if(!get2){
rasterImage(readPNG(D)
,X-SIZE*(vec[2]-vec[1])/2
,Y-SIZE*(vec[4]-vec[3])/2
,X+SIZE*(vec[2]-vec[1])/2
,Y+SIZE*(vec[4]-vec[3])/2
)
}else{
rasterImage(readPNG(D)
,X-SIZE*(vec[2]-vec[1])/2
,Y-SIZE*(vec[4]-vec[3])/2
,X+SIZE*(vec[2]-vec[1])/4
,Y+SIZE*(vec[4]-vec[3])/2
)
rasterImage(readPNG(D)
,X-SIZE*(vec[2]-vec[1])/4
,Y-SIZE*(vec[4]-vec[3])/2
,X+SIZE*(vec[2]-vec[1])/2
,Y+SIZE*(vec[4]-vec[3])/2
)
}
}else{
stop("Package png and RCurl are required")
}
}
deldirでコンビニ位置データのボロノイ分割を行う。
この記事はRAdventCalendar2013に参加しています。
この記事は4日目になります。
これがはじめてというわけではないですがRのdeldirパッケージを使ってみました。
パッケージdeldirはドロネー(Delaunay)分割とディリクレ(Dirichlet)分割(ボロノイ分割、ティーセン分割)に関連する処理を含むパッケージです。
パッケージdeldirの胆になるのが関数delirです。分割に当たってはLee-Schaterアルゴリズムを使用しているようです。
deldir関数の解説
deldir(x, y, dpl=NULL, rw=NULL, eps=1e-09, sort=TRUE, plotit=FALSE,
digits=6, z=NULL, zdum=NULL, suppressMsge=FALSE, ...)
引数
x,y:分割したい座標の入力点
dpl:リスト
rw:分割を行う境界領域を指定する。デフォルトだと入力点の端に対してプラス10%になる。
sort:TRUEがデフォルト。TRUEにセットするとデータが分割前にソートされ計算が効率的になる。
FALSEだと追加点が最後に付加されるので、デバッグ用に設定するための変数である。
plotit:plotを行うか否かのフラグである。
digists:数値の丸め桁数、デフォルトは6桁である。
z:各入力点に対応する重みの値である。
zdum:各ダミー入力点に対応する重みの値である。
返り値
delsgs:ドロネー三角形エッジリストを表すデータフレームである。エッジの座標点(x1,y1,x2,y2)が入力され、各ドロネー点のIDがそれに続く。
dirsgs:ディリクレ領域(ボロノイ領域)エッジリストを表すデータフレームである。エッジの座標点(x1,y1,x2,y2)が入力され、各ドロネー点のIDがそれに続く。
7番目の点はエッジの両端が境界領域と接しているかどうかを表す。
summary:分割されるデータ点とダミー点の情報を含むデータフレーム
-x,y(,z):点とその重みの位置情報
-n.tri:当該点におけるドロネー三角形の数
-del.area:当該点におけるドロネー三角形の面積の3分の1
-del.wts:del.areaを比率に変化させたもの。
-n.tside:ディリクレタイルの他のディリクレタイルとの接触数
-nbpt:ディリクレタイルの境界領域との接触数
-dir.area:ディリクレタイルの面積
-dir.wts:dir.areaの面積を比率に変化させたもの。
n.data:分割されたデータ点、重複は除く。
n.dum:分割されたデータ点、重複点は除く。
del.area:三角に分割された点の凸包の面積、del.areaの合計と一致。
dir.area:境界領域によって囲まれている面積、dir.areaの合計と一致。
rw:正方形領域の枠。
パッケージdeldirその他の関数
tile.list():deldirを受け取って、ボロノイ分割されたタイルを返す。
plot.deldir():deldirを受け取って、プロットする。
以下では、前回の記事でも用いたコンビニデータから東京のコンビニ6009店*1をボロノイ分割してポリゴンをシェープファイルに出力するコードを記載します。
コード
library(deldir)
library(maptools)
#データは事前に準備している。
#> head(df)
#storename storetype x y
#1 セブンイレブン 世田谷中央病院店 セブンイレブン 139.6511 35.64166
#2 ファミリーマート 池袋二丁目店 ファミリーマート 139.7090 35.73495
#3 みんなのイチバ 足立江北2丁目店 その他 139.7637 35.76921
#ボロノイ分割を行う。rwオプションを指定し外枠を拡張させない。
dd<-deldir(df$x, df$y, rw=c(min(df$x),max(df$x),min(df$y),max(df$y)))
#タイルを取得する。
w<-tile.list(dd)
#ポリゴンベクターの初期化
polys<-vector(mode='list', length=length(w))
for (i in seq(along=polys)) {
#ボロノイ点を取得している
pcrds<-cbind(w[[i]]$x, w[[i]]$y)
#閉路化してポリゴン座標に変更する。
pcrds<-rbind(pcrds, pcrds[1,])
#ポリゴンを作成する。
polys[[i]]<-Polygons(list(Polygon(pcrds)), ID=as.character(i))
}
#スパシャルポリゴンオブジェクトの作成
SP<-SpatialPolygons(polys)
#ボロノイ分割したデータをSpatialDataFrameクラスに変更する。
voronoi<-SpatialPolygonsDataFrame(SP,data=data.frame(x=df$x,y=df$y,storename=df$storename, storetype=df$storetype,
row.names=sapply(slot(SP, 'polygons'),function(x) slot(x, 'ID'))))
#shpファイルにして出力する。
writeSpatialShape(voronoi, fn="vornoi-combini")
結果
以下は結果のシェープファイルをQGISで表示させたものです(赤がセブンイレブン、青がローソン、緑がファミリーマート、オレンジがサークルK・サンクス、その他が紫)。
2013年も一月を切りました。皆様よいクリスマス、年越しを。
*1:データ数は6182だったがジオコードの結果、同一座標となった店舗からはランダムに選出した。
Rのspdepのパッケージを使って、コンビニのドミナント出店戦略を定量化する
こんにちは、集計野郎マクガイバーです。
コンビニの出店戦略ではセブンイレブンのドミナント戦略が有名で公式ホームページにも記載してあるくらいです。
しかし、その実態に関してはどの程度そうであるのかといったような厳密な検証情報はなく、特定都市における出店数を比較したようなものや、単に四国にセブンイレブンが出店していないという情報を根拠にしたものが多数見られます。
関西ではローソンがドミナントしているのはいうまでもないし、特定の地域を抜き出して一般的な結論を持ち出すのは問題に思われます。
幸いなことに去年集めたものですが、データが手元にあるので、今日はこのドミナント出店戦略を数値化、可視化してみます。
(以後、ですます調ではなくなります。)
ロウデータで見る各コンビニの出店戦略
ここでは、ロウデータを地図上にプロットして各コンビニチェーンの出店戦略がどのようになっているかを見てみる。
赤:セブンイレブン、青:ローソン、緑:ファミリーマート、オレンジ:サークルK、紫:その他
以下の図は東京都内の中で各チェーンの出店戦略を最も顕著に表していると考えられる箇所を抜粋したもである。

青のローソンが多く見えるが、これは色の見えやすさも要因であることに注意する。
ここから見てとれる出店戦略の仮説は以下の通りである。
セブンイレブン:東京の中心部からやや離れた地域に店舗が多く、電車の沿線ではないところにも出店している。
サークルK:出店地域は狭く傾向は読み取りにくいが、セブンイレブンと同様の出店傾向が見られる。
ローソン:都心を中心に幅広く出店している。沿線上の出店傾向が強い。
ファミリーマート:都心を重点的に出店している。
こうしてみてみると沿線上の出店傾向があるローソンもセブンイレブンと変わらないドミナント戦略のように見える。
ドミナンスを可視化する
続いて、各コンビニチェーンのドミナンスを可視化する。
ここでは、日本地図においてコンビニが存在している領域を切り出したバウンディングボックスを
一辺の最大セル数が300になるように正六角形で区切り、当該セル内における最も店舗数の多いコンビニチェーンを当該チェーンの支配している地域として色を塗りわける。
これを支配地域と呼ぶ。ただし、同一セル内で最も多い出店数が複数チェーンでタイであった場合はランダムにその中から選ぶものとする。
上記の図は後出しではあるが、各コンビニチェーンの出店戦略を最も表していると考えられる地図である。
中国地方から東海地方までを切り出している。色は下記の通りである。
赤:セブンイレブン、青:ローソン、緑:ファミリーマート、オレンジ:サークルK、紫:その他
各チェーンの特徴をまとめると以下のように読み解ける。
セブンイレブン:典型的なドミナント戦略、中国地方や滋賀などに重点的に出店しているが、他の地方には見られない。
サークルK:東海地方に重点的に出店しており、ほとんどの領域を支配している。
ローソン:足元である関西地方の店舗を支配している他に、比較的人口が少ないと思われる山間部や地方にも幅広く出店している。
ファミリーマート:ドミナントしている地域は少ないが、淡路島やその他人口が少ないと思われる地方に所々見られるように幅広く出店している。
またこの地図にはないが、東京の都心部、山手線圏内はファミリーマートが支配している(4セル)。
ちなみに日本全国で見ると以下のような感じになる。
ドミナント出店を定量化する。
可視化では仮説のレベルを出ないため、ドミナント出店をしているかしていないかを表す指標を作成する。
定量化するにあたって、その統計量として以下の3つの条件が必要である。
- ドミナント性・・・ある特定の地域を支配していること。これは先ほどの六角形グリッドのデータを用いることで満たす事が出来る。
- 空間的自己相関性・・・支配地域が空間的に隣接している数が多いこと。支配地域が隣接していること。
- 支配地域の数に対する非依存性・・・隣接数は支配地域が多いと高くなるので、店舗数の少ないチェーンには不利であるため。
この条件を満たす空間的自己相関の統計量として、今回はカテゴリカルな空間的自己相関のjoint-countの検定に用いるZスコアが使えそうなので、これを用いることにした。
(これ以外に上記の条件を満たし、ドミナント戦略を定量化できるよりよい指標は思い浮かばなかった。)
このスコアは帰無仮説(各チェーンにおける支配地域数を変えずにランダムにセルを配置した場合)におけるチェーンの支配地域の隣接数と実際のデータの隣接数を
標準偏差を踏まえた形で比較するものである。Rのspdepのjointcount.multiを用いることで算出できる。
結果
結果は以下の通りである。

Zスコアでは、サークルK・サンクスが最も高い数値を示している事がわかった。
実際にはサークルK・サンクスもセブンイレブンと変わらない程度の高いドミナント戦略をとっている事がわかった。
また、これと比べるとファミリーマートやローソンは幅広く出店する戦略を採択している。
関西を足元に持つローソンがドミナンス指標ではファミリーマートよりやや優位にでる。
また、店舗数比率/支配地域比率のインデックス(より少ない店舗で地域を支配しているという指標)と空間的自己相関性を表すZスコアが比例する形になった。
結論
セブンイレブンとサークルK・サンクスが最も高いドミナント戦略をしていたチェーンに群となる。
その後、ローソン、ファミリーマートと続いた。
最後に
地図はQJIS使って作っています。ドロネー分割したり、人口や沿線とその乗降客数の情報等も使うと他のチェーンの戦略がわかって面白そう。
ちなみに今回この分析をしているのは、コンビニの出店戦略に興味があったからではなく、別の技術的な検証を行うためでした。
それについてはまた時間があれば、ブログを更新したいと思います。
追記:すいません、結論内部で間違った記述をしていた箇所があるため、本文を修正しました。
arulesSequenceをWindowsRStudioから呼び出せるように修正するパッチ
わんばんこ、年末も近づいてきてアソシエーション真っ盛りの季節ですね!
さて、RのarulesSequenceパッケージのcspade関数をWindows7でRStudioから呼び出そうとしたところ、エラーになる事象が発生。
同様の事象は海外でも報告されている。(以下のリンクを参照)
http://grokbase.com/t/r/r-help/124gp26w8r/r-system2-and-cspade-on-windows
https://stat.ethz.ch/pipermail/r-help/2012-April/308802.html
特徴的なのは、RStudioから呼び出そうするとエラーになるが、普通のRConsoleから呼び出す時には動くこと。
で、原因調べてみるとcspade内でsystem2コマンドで実行した結果の標準出力の出力先をstdoutオプションで指定できるはずだが、stdout引数で指定された出力先にリダイレクトされていないのが原因の模様。このため、本来Rのテンプディレクトリに出力されているはずの標準出力結果をリダイレクトしたファイルが存在しないと怒られる。
> s<-cspade(zaki, parameter=list(support=0.5), control = list(verbose = TRUE)) parameter specification: support : 0.5 maxsize : 10 maxlen : 10 algorithmic control: bfstype : FALSE verbose : TRUE summary : FALSE preprocessing ... 1 partition(s), 0 MB [0.02s] mining transactions ... NA MB [0.01s] reading sequences ... 以下にエラー file(con, "r") : コネクションを開くことができません 追加情報: 警告メッセージ: In file(con, "r") : ファイル 'C:\Users\******\AppData\Local\Temp\Rtmp2VKdgt\cspade16503e135e49.out' を開くことができません: No such file or directory
でもって、さらに調べてみると、cspadeコードの上部で、この問題に対応できるようにWindowsの場合のsystem2関数を上書きしている。
ただし、GUIがRStudioの場合を考慮していない。このため、RGuiであれば正しく実行できるが、RStudioだと実行できないということになる。
そこで、この問題を解決するためのアドホックパッチを作成した。
下記のコードを実行すれば解決すると思われる。ただし、cspadeではなく、arulesSequencesのcspadeを呼び出すこと。
ポイントは#change modified function's environmentで修正した関数mod.cspadeに対して環境を設定しているところ。
これをしないとcspade内部で.formatSPというエクスポートされていない関数が呼び出せず落ちてしまうので要注意。
> s<-arulesSequences::cspade(zaki, parameter=list(support=0.5), control = list(verbose = TRUE)) parameter specification: 以下にエラー cat(.formatSP(parameter), sep = "\n") : 関数 ".formatSP" を見つけることができませんでした
パッチ
library(arulesSequences)
#modified cspade
mod.cspade<-function (data, parameter = NULL, control = NULL, tmpdir = tempdir())
{
#fix only here
if (.Platform$OS == "windows" && .Platform$GUI %in% c("Rgui", "RStudio"))
system2 <- function(command, args = character(), stdout = "",
...) {
if (is.character(stdout) && nzchar(stdout)) {
args <- c(args, ">", stdout)
stdout <- NULL
}
args <- c("/c", shQuote(command), args)
command <- Sys.getenv("COMSPEC")
if (!nzchar(command))
stop("environment variable 'COMSPEC' not set")
base::system2(command, args = args, stdout = stdout,
...)
}
if (!inherits(data, "transactions"))
stop("'data' not of class transactions")
if (!all(c("sequenceID", "eventID") %in% names(transactionInfo(data))))
stop("slot transactionInfo: missing 'sequenceID' or 'eventID'")
if (!all(dim(data)))
return(new("sequences"))
parameter <- as(parameter, "SPparameter")
control <- as(control, "SPcontrol")
if (control@verbose) {
t1 <- proc.time()
cat("\nparameter specification:\n")
cat(.formatSP(parameter), sep = "\n")
cat("\nalgorithmic control:\n")
cat(.formatSP(control), sep = "\n")
cat("\npreprocessing ...")
}
exe <- "bin"
if (.Platform$r_arch != "")
exe <- file.path(exe, .Platform$r_arch)
exe <- system.file(exe, package = "arulesSequences")
file <- tempfile(pattern = "cspade", tmpdir)
on.exit(unlink(paste(file, "*", sep = ".")))
opt <- ""
nop <- ceiling((dim(data)[1] + 2 * length(data@data@i)) *
.Machine$sizeof.long/4^10/5)
if (length(control@memsize)) {
opt <- paste("-m", control@memsize)
nop <- ceiling(nop * 32/control@memsize)
}
if (length(control@numpart)) {
if (control@numpart < nop)
warning("'numpart' less than recommended")
nop <- control@numpart
}
out <- paste(file, "stdout", sep = ".")
if (FALSE) {
asc <- paste(file, "asc", sep = ".")
write_cspade(data, con = asc)
if (system2(file.path(exe, "makebin"), args = c(asc,
paste(file, "data", sep = "."))) || system2(file.path(exe,
"getconf"), args = c("-i", file, "-o", file), stdout = out))
stop("system invocation failed")
file.append("summary.out", out)
}
else makebin(data, file)
if (system2(file.path(exe, "exttpose"), args = c("-i", file,
"-o", file, "-p", nop, opt, "-l -x -s", parameter@support),
stdout = out))
stop("system invocation failed")
file.append("summary.out", out)
if (length(parameter@maxsize))
opt <- paste(opt, "-Z", parameter@maxsize, collapse = "")
if (length(parameter@maxlen))
opt <- paste(opt, "-z", parameter@maxlen, collapse = "")
if (length(parameter@mingap))
opt <- paste(opt, "-l", parameter@mingap, collapse = "")
if (length(parameter@maxgap))
opt <- paste(opt, "-u", parameter@maxgap, collapse = "")
if (length(parameter@maxwin))
opt <- paste(opt, "-w", parameter@maxwin, collapse = "")
if (!length(control@bfstype) || !control@bfstype)
opt <- paste(opt, "-r", collapse = "")
if (control@verbose) {
t2 <- proc.time()
du <- sum(file.info(list.files(path = dirname(file),
pattern = basename(file), full.names = TRUE))$size)
cat(paste("", nop, "partition(s),", round(du/4^10, digits = 2),
"MB"))
cat(paste(" [", format((t2 - t1)[3], digits = 2, format = "f"),
"s]", sep = ""))
cat("\nmining transactions ...")
}
out <- paste(file, "out", sep = ".")
if (system2(file.path(exe, "spade"), args = c("-i", file,
"-s", parameter@support, opt, "-e", nop, "-o"), stdout = out))
stop("system invocation failed")
if (control@verbose) {
t3 <- proc.time()
du <- file.info(out)$size
cat(paste("", round(du/4^10, digits = 2), "MB"))
cat(paste(" [", format((t3 - t2)[3], digits = 2, format = "f"),
"s]", sep = ""))
cat("\nreading sequences ...")
}
out <- read_spade(con = out, labels = itemLabels(data))
out@info <- c(data = match.call()$data, ntransactions = length(data),
out@info, support = parameter@support)
if (control@verbose) {
t4 <- proc.time()
cat(paste(" [", format((t4 - t3)[3], digits = 2, format = "f"),
"s]", sep = ""))
cat("\n\ntotal elapsed time: ", (t4 - t1)[3], "s\n",
sep = "")
}
if (!control@summary)
unlink("summary.out")
out
}
#change modified function's environment
environment(mod.cspade)<-environment(cspade)
#unlock and assign
unlockBinding("cspade", getNamespace("arulesSequences"))
assign("cspade", mod.cspade, getNamespace("arulesSequences"))
lockBinding("cspade", getNamespace("arulesSequences"))
#doing cspade(note:cpade itself does not change.try to call cspade not arulesSequences::cspade)
data(zaki)
s<-arulesSequences::cspade(zaki, parameter=list(support=0.5), control = list(verbose = TRUE))
Windows8にrstanをインストールする。
こんにちわ。BUGSにはまってしまって大変なアナリストです。
どうにもこうにもWINBUGSが遅くていや遅すぎて使えないので、今日はRからstanを叩くrstanをインストールしてみたいと思います。
環境はWindows8 64bitです。
1.Rtoolsをインストール
以下のサイトからRtoolsをインストールしてc++のコンパイルができるようにしておきます。
http://cran.r-project.org/bin/windows/Rtools/
適当に次へ次へをクリックしておけば問題ないです。
今回はRtools31.exeをインストールします。
途中環境変数の設定があったりしますが、そのままで問題ないです。
【追記】この時、Windowsでは、PATHは先頭にあるものを優先して実行するのですでにコンパイラーなどをインストールしている場合はPATHの先頭にRtoolsの実行ファイルが含まれているフォルダが存在するのを確認してください。
2.rstanのインストール
次のコードを実行します。
#必要なパッケージのインストール
install.packages('inline')
install.packages('Rcpp')
#hello worldと表示されたら、インストールに問題ない。
library(inline)
library(Rcpp)
src <- '
std::vector<std::string> s;
s.push_back("hello");
s.push_back("world");
return Rcpp::wrap(s);
'
hellofun <- cxxfunction(body = src, includes = '', plugin = 'Rcpp', verbose = FALSE)
cat(hellofun(), '\n')
#rstan自体のインストール
Sys.setenv(R_MAKEVARS_USER='')
options(repos = c(getOption("repos"), rstan = "http://wiki.stan.googlecode.com/git/R"))
install.packages('rstan', type = 'source')
library(rstan)
Rのflexmixパッケージで混合分布モデルによるクラスタ分析を行う。
Rで混合分布クラスタリングを行うときに有名なパッケージとしてflexmixが存在します。この記事ではflexmixの簡単な使い方を解説します。
flexmix自体は潜在クラス回帰を行うパッケージなのですが、混合分布クラスタリングを行うことも出来ます。
flexmixはRのglmクラスを用いてモデルを表現出来るため、他のパッケージに比べて柔軟なモデリングが可能というメリットがあります。
そもそも、混合分布クラスタリングとはなんぞやという人は以下の本文を参考にしてください。
1.モデルベースのクラスタリングとは
クラスタリングは代表的なものとして、以下の3つの方法が存在します。
おそらくk-meansと階層的クラスタ分析はみなさんご存知でしょう。
| 分類 | メリット・デメリット | 手法 |
|---|---|---|
| 階層的手法 | +データを樹形図として表現可能 ‐データ数が多いと、樹形図として表現できないのでデータ数が絞られる。 |
階層的クラスタ分析 |
| 分割的手法 | +代表点を入手可能 ‐代表点以外のデータの関係性やモデルに関する知識が得られない。 |
k-means PAM |
| モデルベース手法 | +データの発生メカニズムを知ることが出来る。 ‐事前にある程度のデータ発生メカニズムについて知る必要がある。 |
混合分布モデル |
混合分布を用いたクラスタリングはクラスタリングをする際にモデル構築するため、モデルベースのクラスタリングに属します。
階層的クラスタリングのようにデータ間距離の設定などがいらず、すべて確率という尺度に直した上で、クラスタリングできる。
またデータ発生メカニズムを知ることができるため成功すれば、非常に強力な手法であると言えます。
2.混合分布を用いたクラスタリング
混合分布を用いたクラスタリングについては@sleipnir002さんが、TokyoRで発表された以下のスライドを参考にしてください。
ちなみにこれは以下の本を参考にしているとのことなので、こちらの本の第4章も参考にしてください。

- 作者: 金森敬文,竹之内高志,村田昇,金明哲
- 出版社/メーカー: 共立出版
- 発売日: 2009/10/23
- メディア: 単行本
- 購入: 5人 クリック: 61回
- この商品を含むブログ (11件) を見る
3.混合分布一般によるクラスタリングへ
上記で紹介している手法はパッケージMClustを用いているため、データ発生メカニズムとしては正規分布しか用いることができません。
私は小売向けのアナリストですが、回数のデータなんかはポワソン分布だし、性別比とかは二項分布だし、正規分布だけだと色々と厳しいのが実情です。
そこで、もっと柔軟にモデルを表現する必要があります。
4.Rでの実行例
flexmixは潜在クラス回帰を行うためのパッケージですが、入力するglmのモデル式の形を工夫することで、混合分布クラスタリングを行うことが出来ます。
flexmixを実行して、クラスタ分析を行う例を用いて解説しましょう。
library(flexmix) #A.ポワソン分布×2と正規分布変数のデータ(y1,y2,y3)を作成 #クラスタ数は3(100, 100, 20) y1<-c(rpois(100, 3), rpois(100, 8), rpois(20, 6)) y2<-c(rpois(100, 10), rpois(100, 0.5), rpois(20, 6)) y3<-c(rnorm(100, mean=200, 15), rnorm(100, mean=200, 10),rnorm(20, mean=200, 10)) class<-c(rep(1, 100), rep(2, 100), rep(3, 20)) data<-data.frame(y1,y2,y3, class) rm(y1, y2, y3, class) #y1 barplot(table(data[, c(4,1)])) #y2 barplot(table(data[, c(4,2)])) #y3 plot(data[, 3], col=data[,4]) #B.混合分布モデルのコンポーネントをそれぞれ作成 Model_1<-FLXMRglm(y1 ~ 1,family="poisson") Model_2<-FLXMRglm(y2 ~ 1,family="poisson") Model_3<-FLXMRglm(y3 ~ 1,family="gaussian") #C.EMアルゴリズムの実施 mp<-flexmix(. ~ ., data=data, k=3, model=list(Model_1, Model_2, Model_3), control=list(verbose=1, nrep=5)) # Classification: weighted # 1 Log-likelihood : -2405.1507 # 2 Log-likelihood : -2173.0186 # 3 Log-likelihood : -1936.5001 # 4 Log-likelihood : -1911.7593 # 5 Log-likelihood : -1905.8872 # 6 Log-likelihood : -1903.8647 # 7 Log-likelihood : -1902.4038 # 8 Log-likelihood : -1901.2357 # 9 Log-likelihood : -1900.3630 # 10 Log-likelihood : -1899.6985 # 11 Log-likelihood : -1899.1289 # 12 Log-likelihood : -1898.5728 # 13 Log-likelihood : -1897.9860 # 14 Log-likelihood : -1897.3486 # 15 Log-likelihood : -1896.6569 # 16 Log-likelihood : -1895.9206 # 17 Log-likelihood : -1895.1609 # 18 Log-likelihood : -1894.4037 # 19 Log-likelihood : -1893.6678 # 20 Log-likelihood : -1892.9590 # 21 Log-likelihood : -1892.2749 # 22 Log-likelihood : -1891.6134 # 23 Log-likelihood : -1890.9800 # 24 Log-likelihood : -1890.3919 # 25 Log-likelihood : -1889.8763 # 26 Log-likelihood : -1889.4597 # 27 Log-likelihood : -1889.1529 # 28 Log-likelihood : -1888.9460 # 29 Log-likelihood : -1888.8157 # 30 Log-likelihood : -1888.7373 # 31 Log-likelihood : -1888.6915 # 32 Log-likelihood : -1888.6652 # 33 Log-likelihood : -1888.6502 # 34 Log-likelihood : -1888.6417 # 35 Log-likelihood : -1888.6369 # 36 Log-likelihood : -1888.6342 # 37 Log-likelihood : -1888.6327 # converged #D.サマリを表示する。 summary(mp) # Call: # flexmix(formula = . ~ ., data = data, k = 3, model = list(Model_1, Model_2, Model_3), control = list(verbose = 1, # nrep = 5)) # # prior size post>0 ratio # Comp.1 0.436 94 135 0.696 # Comp.2 0.445 100 117 0.855 # Comp.3 0.119 26 194 0.134 # # 'log Lik.' -1888.633 (df=14) # AIC: 3805.265 BIC: 3852.776 #E.パラメータを取得する。 parameters(mp) # [[1]] # Comp.1.coef.(Intercept) Comp.2.coef.(Intercept) Comp.3.coef.(Intercept) # 0.9826601 2.1248895 1.7702220 # # [[2]] # Comp.1.coef.(Intercept) Comp.2.coef.(Intercept) Comp.3.coef.(Intercept) # 2.3623702 -0.6625998 1.6267241 # # [[3]] # Comp.1 Comp.2 Comp.3 # coef.(Intercept) 200.23493 202.094819 200.380162 # sigma 15.13504 9.412856 5.797705 #F.交差表の作成 table(clusters(mp), data$class) # # 1 2 3 # 1 91 0 3 # 2 0 98 2 # 3 9 2 15 #G.結果のプロット plot(mp)
A.ポワソン分布×2と正規分布変数のデータ(y1,y2,y3)を作成
今回、y1、y2、y3という3つの変数をクラスタ分析にかけます。
変数は以下の分布に従うとします。
| 変数名 | 分布 |
|---|---|
| y1 | ポワソン分布 |
| y2 | ポワソン分布 |
| y3 | 正規分布 |
例えば、小売の顧客分類で例えると、y1は来店回数、y2商品の購入数、y3は利用金額(対数正規に従う)対数正規に従う利用金額の平均が200ってどうなの・・・とか言わないでください。といった例が考えうるでしょうか。
また、このデータには3つのクラス(クラスタ)がいて、それぞれ以下のようなパラメータを持っているとします。
| 変数名 | n(サイズ) | y1~λ | y2~λ | y3~μ | y3~σ |
|---|---|---|---|---|---|
| クラス1 | 100 | 3 | 10 | 200 | 15 |
| クラス2 | 100 | 8 | 0.5 | 200 | 10 |
| クラス3 | 20 | 6 | 6 | 200 | 10 |
クラス別のy1のヒストグラムが以下の図です。

クラス別のy2のヒストグラムが以下の図です。
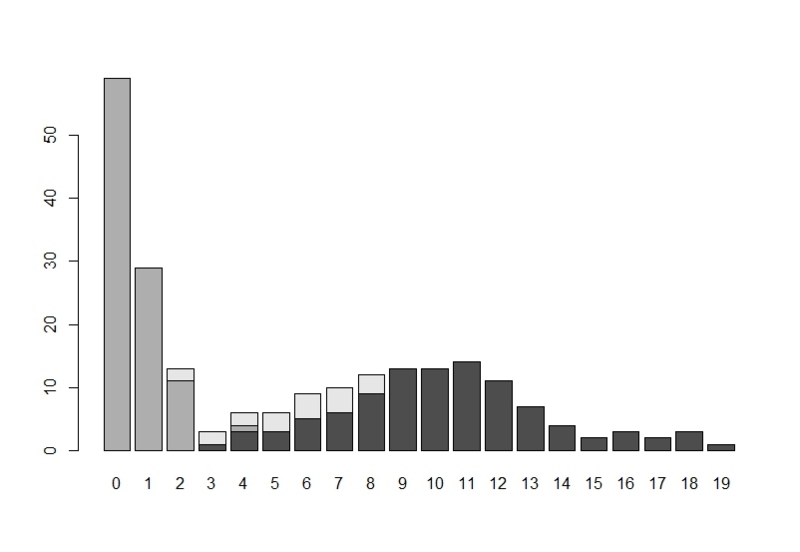
y3の値を各個体別にプロットしたが以下の図です。
(左からクラス1、2、3と続いています。)

B.混合分布モデルのコンポーネントをそれぞれ作成
ここでは、flexmixに入力するモデル式をglm形式で、作成しています。
ポイントは回帰式の説明変数が定数で、被説明変数が各変数yに対応しているということです。
C.EMアルゴリズムの実施
flexmix関数でEMアルゴリズムを実施して、混合分布モデルによるクラスタリングを行います。
kは想定しているコンポーネントの数を、modelには先ほど作成したglmモデルをリスト化して代入します。
controlはアルゴリズムのチューニング用です。
convergedと表示されていれば収束しています。
D.サマリを表示する。
summaryで実行結果のサマリを閲覧することが出来ます。
E.パラメータを取得する。
parametersで推定された各クラスタ毎のモデルのパラメータを見ることができます。
リストの1から順に、y1(Model_1)、y2(Model_2)、y3(Model_3)のパラメータを返しています。
(ポワソン分布のパラメータは対数です。)
実際のクラス別のパラメータ
| 変数名 | n(サイズ) | y1~λ | y2~λ | y3~μ | y3~σ |
|---|---|---|---|---|---|
| クラス1 | 100 | 3 | 10 | 200 | 15 |
| クラス2 | 100 | 8 | 0.5 | 200 | 10 |
| クラス3 | 20 | 6 | 6 | 200 | 10 |
推定されたクラス別のパラメータ
| 変数名 | n(サイズ) | y1~λ | y2~λ | y3~μ | y3~σ |
|---|---|---|---|---|---|
| クラス1 | 94 | 2.671553 | 10.61608 | 200.23493 | 15.13504 |
| クラス2 | 100 | 8.371972 | 0.5155094 | 202.094819 | 9.412856 |
| クラス3 | 26 | 5.872157 | 5.087182 | 200.380162 | 5.797705 |
F.交差表の作成
clusters(mp)でデータの属するクラスの値を取得出来ます。
これを実際のdataの属するクラスと比較しています、クラス3に多少の誤差はあるものの、分類できているようです。
G.結果のプロット
結果をplot関数に投げると、各クラスのクラス事後確率のヒストグラムが表示されます。

というわけで、flexmixの簡単な使い方を紹介ました。
細かなパラメータ設定についてはマニュアルを参照してください。
また、ガイドが結構丁寧なので、この記事を読まれて興味を持たれた方はぜひこちらを参照してください。
参考
FlexMix: A General Framework for Finite Mixture Models and Latent Class Regression in R
FlexMix Version 2: Finite Mixtures with Concomitant Variables and Varying and Constant Parameters
IT苦手な人のためのWindows+gitでRStudioによるバージョン管理入門
『何を求め、どこへ行くのか』〜こんにちはR界の若頭です。
WindowsでRStudioを使ってRのコードを書いている人って、多いと思います。IT苦手だけど、データマイニングしたいという人に多いんじゃないでしょうか。で、RStudioにいつからだったかバージョン管理機能がついていて、「同じようなファイルがフォルダに混在しているしバージョン管理使ってみたいけどIT苦手だしよくわかんねーよ。日本語の手順解説ブログねーし。」というIT苦手な自分、いやあなたのために、今日はWindowsのRStudioによるバージョン管理機能をgitで有効化した時の手順を記しておきます。
私が設定した環境を記載しておきます。
Windows7(64bit)
RStudio0.97
git version 1.8.0.msysgit.0
大きな手順は以下のとおりです。
1.gitのインストール
2.gitのセットアップ
3.RStudioでの新規プロジェクトの作成
4.コードのバージョン管理
5.バージョンを戻す方法
1.gitのインストール
gitのインストールは他に詳しくてわかりやすい記事が沢山あるので、ここでは省略します。
インストール方法はこちら。
Windows で Git の環境設定 (msysgit, TortoiseGit)
Gitとは何かについてはこちらがわかりやすいです。
サルでもわかるGit入門
2.gitのセットアップ
インストールが終わるとやることは2つです(これは先のサイトでも解説されています。)。
1.git.exeの入ったフォルダ名を環境変数PATHに追加しておく。
2.コマンドプロンプトより、以下のコマンドをおまじないとして打つ。
git config --global user.email "you@example.com"
git config --global user.name "Your Name"
gitに貴方自身の情報を認識させるコマンドだと思えば大丈夫です。
別にメールを送ったりするわけではないので、実在するメールアドレスや名前を入力する必要はありません。
3.RStudioでの新規プロジェクトの作成
RStudio上部のProjectメニューより、新規プロジェクトを作成します。

Projectメニューより、Project Optionを選択します。

Git/SVNより、Version control systemをGitにします。

これで設定完了です。RStudioのプロジェクトのフォルダがGitを用いてバージョン管理されていることになります。
4.コードのバージョン管理
ここでは、test3.Rというファイルをコミットして、バージョン1に更新してみます。
画面右上Gitタブの"Staged"にチェック、"Commit"を押す。

Commit messageにコメントを代入して、"Commit"を押す。

これでtest3.Rがレポジトリ上に保存されています。
複数回繰り返せばバージョンが上がっていくことになります。
5.バージョンを戻す方法
書いたソースコードを昔のバージョンに戻すには、以下の2つの方法があります。
1.現在編集中のコードを最終コミットバージョンに戻す。
現在コミット前の編集中コードの変更を最終コミット時に修正するには、修正対象のファイルを選択し、RevertボタンでOKです。

2.それより古いバージョンに戻す。
古いバージョンにさかのぼってバージョンを戻す場合は、以下の手順でOKです。
先程のtest3.Rがバージョン3まで更新されたとします。これを最初のバージョンに戻すには、
画面右上のGitタブより、More→Historyを選択します。

すると、過去のコミット履歴が参照できるので、最初のコミット時のコードを参照します。

View file@******から過去のコードを参照することができます。これを用いて既存のコードを上書きしましょう。

以上です!
2013年3月29日 一部記事を修正しました。






